
機械学習向けのノートブックを前処理や誤差評価とともにパイプライン化してみた
この記事は公開されてから1年以上経過しています。情報が古い可能性がありますので、ご注意ください。
こんちには。
データ事業本部 インテグレーション部 機械学習チームの中村( @nokomoro3 )です。
今回は、前回の以下の記事で試したNotebook JobをSageMaker Pipelinesに組み込んでいきます。
また、パイプライン作成時にSpace用のコンテナイメージとジョブ用のコンテナイメージをなるべく共通化することで、NotebookをSpaceで検証する時とジョブで実行する時で同じものが使えるように考慮していきます。
SageMaker Pipelinesについて
SageMaker Pipelinesとは、機械学習の各フェーズの処理を step という形で表現して、パイプラインを形成するものです。
SageMaker SDKなどを使って構築することができ、以下に公式サンプルがあります。
こちらを使って構築したパイプラインは、SageMaker StudioのIDEからGUIで確認することもできます。
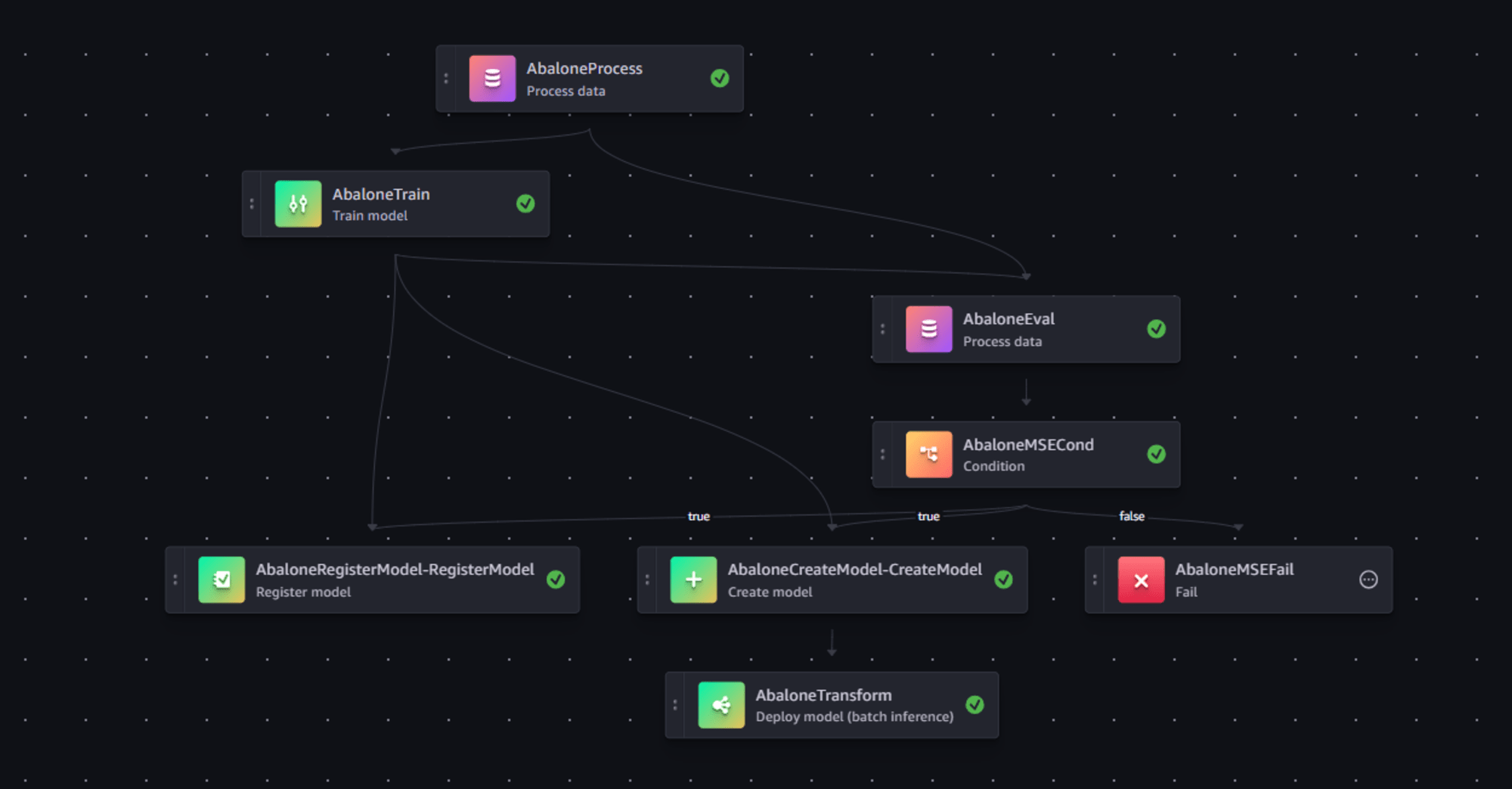
この例では主に以下のフローで処理する例となっています。
- (1) 前処理
- (2) モデル学習
- (3) 学習したモデルをMSE(平均二乗誤差)で誤差を評価
- (4) 誤差が閾値範囲内どうかで条件分岐
- 誤差が想定内:Model Registryへ登録、Model作成、バッチ推論の実行
- 誤差が想定外:FailStepへ移行
パイプラインはGUI上でも一応作成することができます(機能が漏れなく構築できるかは要検証です)。
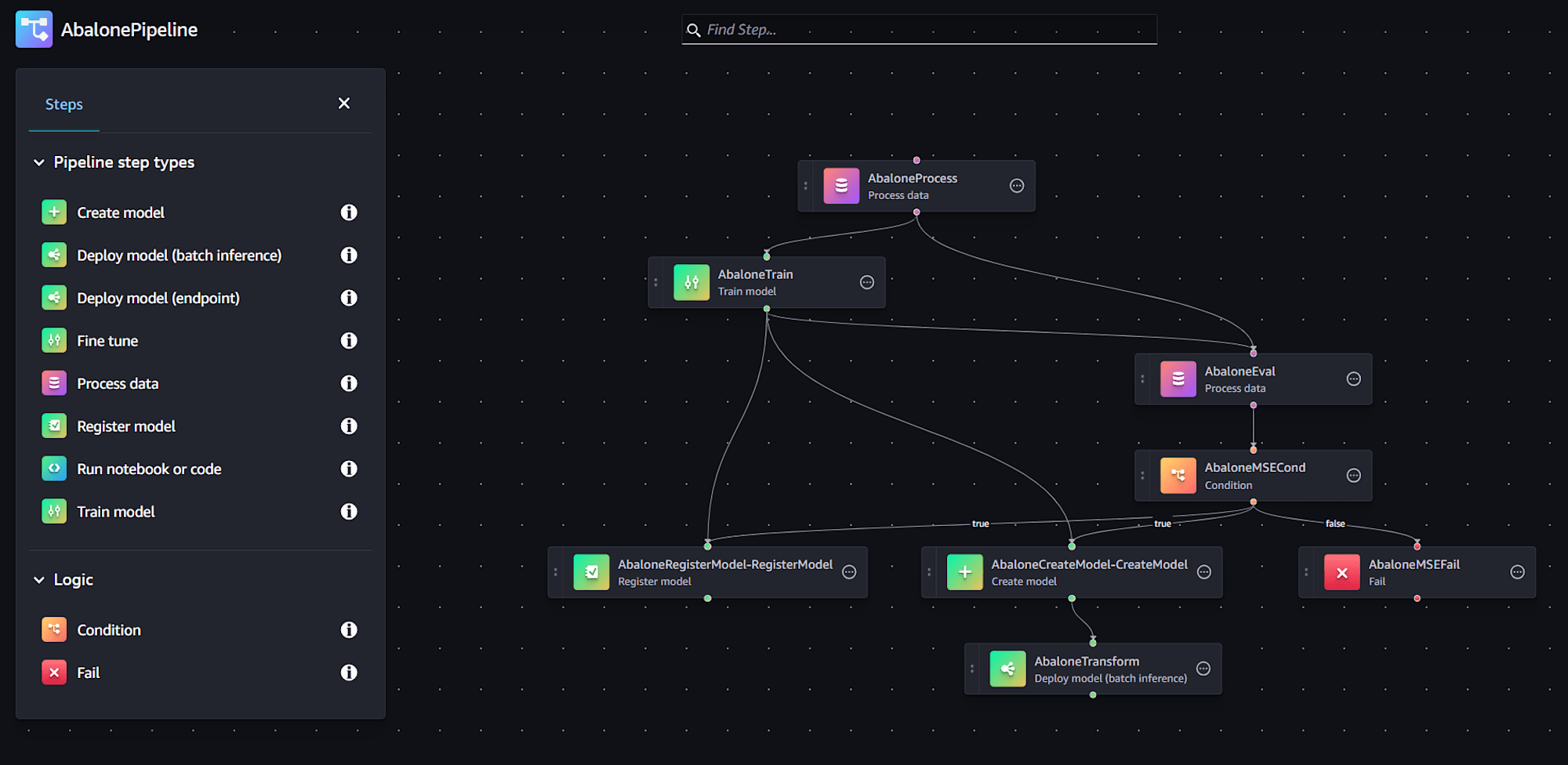
今回はこちらを参考にNotebookJobをパイプラインに組み込み、(1)~(3)までを実行します。
やってみた
コンテナイメージの作成とECRへのアップロード
最初だけdockerやAWS CLIが使えるローカル環境などで作業します。
今回は、Studio上のJupyterLab Spaceと同じコンテナイメージを使って各ジョブ(ProcessingJobやNotebookJob)を実行します。
Space用のイメージとしては以下の sagemaker-distribution がよく使用されています。
NotebookJobはその性質上、Space用と同じイメージで問題ないのですが、ProcessingJobはrootが所有者のフォルダにコンテナ内からアクセスする必要があるので、以下のように sagemaker-distribution を少し変更するような Dockerfile を記述します。
FROM public.ecr.aws/sagemaker/sagemaker-distribution:2.1.0-cpu
# ユーザーをrootに変更
USER root
参考までにSpace用のイメージはUID=1000, GID=100となっている必要があります。
このDockerfileをカスタムイメージとして使用しますので、ECRレポジトリを作成して、
REPOSITORY_NAME="sagemaker-distribution-for-job"
aws ecr create-repository --repository-name ${REPOSITORY_NAME}
docker buildしてECRにpushします。
# imageのビルド
docker build -t ${REPOSITORY_NAME} .
# タグ付け
REGION=$(aws configure get region)
ACCOUNT_ID=$(aws sts get-caller-identity --query 'Account' --output text)
docker tag ${REPOSITORY_NAME}:latest ${ACCOUNT_ID}.dkr.ecr.${REGION}.amazonaws.com/${REPOSITORY_NAME}:latest
# ECRにログイン
aws ecr get-login-password --region ${REGION} | docker login --username AWS --password-stdin ${ACCOUNT_ID}.dkr.ecr.${REGION}.amazonaws.com
# ECRにpush
docker push ${ACCOUNT_ID}.dkr.ecr.${REGION}.amazonaws.com/${REPOSITORY_NAME}:latest
これでコンテナイメージの準備は完了です。作成したイメージのURI
${ACCOUNT_ID}.dkr.ecr.${REGION}.amazonaws.com/${REPOSITORY_NAME}:latest
は控えておきます。
以降は、ローカル環境またはStudio内のJupyterLab SpaceなどのSageMaker SDKを使える環境で作業します。
実行環境のPythonライブラリのバージョン
各モジュールのバージョンは以下です。
pydantic 2.9.0
pydantic-core 2.23.2
sagemaker 2.230.0
sagemaker-core 1.0.3
scikit-learn 1.5.1
まずは各ジョブで必要なファイルを作成していきます。
前処理用のスクリプト作成
train/validation/testにデータセットを分けてCSVとして保存する処理をファイルに記述します。
import numpy as np
import pandas as pd
from sklearn.compose import ColumnTransformer
from sklearn.impute import SimpleImputer
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler, OneHotEncoder
# Since we get a headerless CSV file, we specify the column names here.
feature_columns_names = [
"sex",
"length",
"diameter",
"height",
"whole_weight",
"shucked_weight",
"viscera_weight",
"shell_weight",
]
label_column = "rings"
feature_columns_dtype = {
"sex": str,
"length": np.float64,
"diameter": np.float64,
"height": np.float64,
"whole_weight": np.float64,
"shucked_weight": np.float64,
"viscera_weight": np.float64,
"shell_weight": np.float64,
}
label_column_dtype = {"rings": np.float64}
def merge_two_dicts(x, y):
z = x.copy()
z.update(y)
return z
if __name__ == "__main__":
base_dir = "/opt/ml/processing"
df = pd.read_csv(
f"{base_dir}/input/abalone-dataset.csv",
header=None,
names=feature_columns_names + [label_column],
dtype=merge_two_dicts(feature_columns_dtype, label_column_dtype),
)
numeric_features = list(feature_columns_names)
numeric_features.remove("sex")
numeric_transformer = Pipeline(
steps=[("imputer", SimpleImputer(strategy="median")), ("scaler", StandardScaler())]
)
categorical_features = ["sex"]
categorical_transformer = Pipeline(
steps=[
("imputer", SimpleImputer(strategy="constant", fill_value="missing")),
("onehot", OneHotEncoder(handle_unknown="ignore")),
]
)
preprocess = ColumnTransformer(
transformers=[
("num", numeric_transformer, numeric_features),
("cat", categorical_transformer, categorical_features),
]
)
y = df.pop("rings")
X_pre = preprocess.fit_transform(df)
y_pre = y.to_numpy().reshape(len(y), 1)
X = np.concatenate((y_pre, X_pre), axis=1)
np.random.shuffle(X)
train, validation, test = np.split(X, [int(0.7 * len(X)), int(0.85 * len(X))])
pd.DataFrame(train).to_csv(f"{base_dir}/train/train.csv", header=False, index=False)
pd.DataFrame(validation).to_csv(
f"{base_dir}/validation/validation.csv", header=False, index=False
)
pd.DataFrame(test).to_csv(f"{base_dir}/test/test.csv", header=False, index=False)
この内容を code/preprocessing.py に保存しておきます。
このスクリプトはProcessingJobで実行され、ジョブ作成時にS3と /opt/ml/processing というフォルダを関連付けることで、処理を実現します。
ジョブ用のノートブック作成
ジョブ用のノートブックをまずは作成しておきます。
前回はPyTorchで構成しましたが、今回はサンプルがAbaloneデータセットの回帰問題を、xgboostで解くものとなっているのでそちらに合わせます。
まずは前回の記事でご説明したように入出力の情報は環境変数から取得する必要があるので、以下のようにそれを取得します。
### 入出力情報を環境変数から取得
import os
INPUT_DATASET_TRAIN = os.getenv("INPUT_DATASET_TRAIN")
INPUT_DATASET_VALID = os.getenv("INPUT_DATASET_VALID")
MODEL_OUTPUT_S3_URI = os.getenv("MODEL_OUTPUT_S3_URI")
次にS3からデータを取得して、ファイルとして保存します。
### データ取得
import sagemaker
# セッションの作成
session = sagemaker.Session()
# ダウンロード
session.download_data(
path='train', # ローカルの保存先ディレクトリ
bucket=INPUT_DATASET_TRAIN[5:].split("/")[0],
key_prefix="/".join(INPUT_DATASET_TRAIN[5:].split("/")[1:])
)
session.download_data(
path='validation',
bucket=INPUT_DATASET_VALID[5:].split("/")[0],
key_prefix="/".join(INPUT_DATASET_VALID[5:].split("/")[1:])
xgboostによる学習を行います。
### 学習
import xgboost as xgb
import pandas as pd
# データ読み込み(ヘッダーなしCSVを想定)
train_data = pd.read_csv("train/train.csv", header=None)
validation_data = pd.read_csv("validation/validation.csv", header=None)
# 特徴量とターゲットを分離(0列目がターゲット)
X_train = train_data.iloc[:, 1:] # 2列目以降が特徴量
y_train = train_data.iloc[:, 0] # 1列目がターゲット
X_val = validation_data.iloc[:, 1:]
y_val = validation_data.iloc[:, 0]
# DMatrixの作成
dtrain = xgb.DMatrix(X_train, label=y_train)
dval = xgb.DMatrix(X_val, label=y_val)
# ハイパーパラメータの設定
params = {
'objective': 'reg:linear',
'max_depth': 5,
'eta': 0.2,
'gamma': 4,
'min_child_weight': 6,
'subsample': 0.7
}
# モデルの学習
model = xgb.train(
params=params,
dtrain=dtrain,
num_boost_round=50,
evals=[(dtrain, 'train'), (dval, 'validation')]
)
以降のステップで使用するため、学習したモデルをS3にアップロードします。
### モデル保存
import pickle
# pickleとして保存
with open('model.pkl', 'wb') as f:
pickle.dump(model, f)
import sagemaker
import tarfile
import os
# モデルをtarファイルにパッケージング
with tarfile.open('model.tar.gz', 'w:gz') as tar:
tar.add('model.pkl')
# SageMaker sessionを使ってアップロード
session = sagemaker.Session()
session.upload_data(
path='model.tar.gz',
bucket=MODEL_OUTPUT_S3_URI[5:].split("/")[0],
key_prefix="/".join(MODEL_OUTPUT_S3_URI[5:].split("/")[1:-1])
)
これらを job-abalone-xgboost.ipynb として保存しておきます。
(実際の開発ではこのノートブックをSpaceで検証して、NotebookJobを含んだパイプラインにデプロイしていく流れを想定しています)
誤差評価用のスクリプト作成
前処理と同様、誤差計算もProcessingJobを使いますので、必要なスクリプトを記述します。
import json
import pathlib
import pickle
import tarfile
import os
import joblib
import numpy as np
import pandas as pd
import xgboost
from sklearn.metrics import mean_squared_error
if __name__ == "__main__":
model_path = f"/opt/ml/processing/model/model.tar.gz"
with tarfile.open(model_path) as tar:
tar.extractall(path=".")
model = pickle.load(open("model.pkl", "rb"))
test_path = "/opt/ml/processing/test/test.csv"
df = pd.read_csv(test_path, header=None)
y_test = df.iloc[:, 0].to_numpy()
df.drop(df.columns[0], axis=1, inplace=True)
# モデルの特徴量名を確認
print("Model feature names:", model.feature_names)
# テストデータのカラム名を確認
print("Test data columns:", df.columns)
# カラム名を完全に一致させる
df.columns = model.feature_names
X_test = xgboost.DMatrix(df)
predictions = model.predict(X_test)
mse = mean_squared_error(y_test, predictions)
std = np.std(y_test - predictions)
report_dict = {
"regression_metrics": {
"mse": {"value": mse, "standard_deviation": std},
},
}
output_dir = "/opt/ml/processing/evaluation"
pathlib.Path(output_dir).mkdir(parents=True, exist_ok=True, mode=0o777)
evaluation_path = f"{output_dir}/evaluation.json"
with open(evaluation_path, "w") as f:
f.write(json.dumps(report_dict))
これを code/evaluation.py として保存しておきます。
変数定義
まずは関連する変数を定義します。
roleはStudio内の環境を使うか、ローカル環境から実施するかで異なるので必要に応じて変更ください。
import boto3
import sagemaker
from sagemaker.workflow.pipeline_context import PipelineSession
sagemaker_session = sagemaker.session.Session()
region = sagemaker_session.boto_region_name
# role = sagemaker.get_execution_role() # Studio内でやる場合はこちら
role = "{SageMakerの実行ロール}"
pipeline_session = PipelineSession()
default_bucket = sagemaker_session.default_bucket()
pipeline_sessionはパイプラインの各ステップのジョブ定義時にsagemaker_sessionの代わりに設定することで、そのジョブを実行せずにステップとして構成するためのsessionとなっています。
先ほど控えたコンテナイメージのURIもここで変数に入れておきます。
image_uri="{コンテナイメージのURI}"
print(f"{image_uri=}")
データソースの準備
S3に元となるデータソースをアップロードします。
local_path = "data/abalone-dataset.csv"
s3 = boto3.resource("s3")
s3.Bucket(f"sagemaker-example-files-prod-{region}").download_file(
"datasets/tabular/uci_abalone/abalone.csv", local_path
)
base_uri = f"s3://{default_bucket}/abalone"
input_data_uri = sagemaker.s3.S3Uploader.upload(
local_path=local_path,
desired_s3_uri=base_uri,
)
print(input_data_uri)
この通りに実行すると、default_bucketが s3://sagemaker-{リージョン}-{アカウントID} となりますので、 s3://sagemaker-{リージョン}-{アカウントID}/abalone/abalone-dataset.csv にアップロードされるはずです。
このcsvファイルが、最初の前処理の入力となります。
パイプライン定義
次にパイプラインの定義と各ジョブのステップを作成します。
まずはパイプラインのパラメータを定義します。
from sagemaker.workflow.parameters import (
ParameterInteger,
ParameterString
)
instance_type = ParameterString(
name="TrainingInstanceType",
default_value="ml.m5.xlarge"
)
input_data = ParameterString(
name="InputData",
default_value=input_data_uri
)
model_s3_uri = ParameterString(
name="ModelS3Uri",
default_value=f"s3://{default_bucket}/sample-notebook-job/models/abalone-xgboost/model.tar.gz"
)
次に前処理のステップを作成します。
from sagemaker.processing import ScriptProcessor
from sagemaker.processing import ProcessingInput, ProcessingOutput
from sagemaker.workflow.steps import ProcessingStep
# ProcessingJobの定義
preprocess = ScriptProcessor(
image_uri=image_uri,
command=["python3"],
instance_type=instance_type,
instance_count=1,
base_job_name="abalone-xgboost-preprocess",
role=role,
sagemaker_session=pipeline_session, # ここがpipelinesのポイント
)
# pipeline_sessionなのでrunしてもジョブが動作せず、、、
preprocess_args = preprocess.run(
inputs=[
ProcessingInput(source=input_data, destination="/opt/ml/processing/input"),
],
outputs=[
ProcessingOutput(output_name="train", source="/opt/ml/processing/train"),
ProcessingOutput(output_name="validation", source="/opt/ml/processing/validation"),
ProcessingOutput(output_name="test", source="/opt/ml/processing/test"),
],
code="code/preprocessing.py",
)
# runの戻り値をStepに与えられる
step_preprocess = ProcessingStep(name="abalone-xgboost-preprocess", step_args=preprocess_args)
コメントに記載のとおり、通常はrunととものProcessingJobが実行されるのですが、pipeline_sessionを使うことでジョブをパイプラインのステップとして扱うことができます。
次に学習用のステップとして、NotebookJobのステップを作成します。NotebookJobの入出力は環境変数として渡します。
from sagemaker.workflow.notebook_job_step import NotebookJobStep
step_notebook_job = NotebookJobStep(
notebook_job_name="abalone-xgboost-notebook-job",
input_notebook="./job-abalone-xgboost.ipynb",
image_uri=image_uri,
kernel_name="python3",
instance_type=instance_type,
role=role,
environment_variables={
"INPUT_DATASET_TRAIN": step_preprocess.properties.ProcessingOutputConfig.Outputs["train"].S3Output.S3Uri,
"INPUT_DATASET_VALID": step_preprocess.properties.ProcessingOutputConfig.Outputs["validation"].S3Output.S3Uri,
"MODEL_OUTPUT_S3_URI": model_s3_uri,
}
)
最後に誤差評価用のProcessingJobのステップを作成します。
from sagemaker.processing import ScriptProcessor
from sagemaker.processing import ProcessingInput, ProcessingOutput
from sagemaker.workflow.steps import ProcessingStep
# ProcessingJobの定義
evaluation = ScriptProcessor(
image_uri=image_uri,
command=["python3"],
instance_type=instance_type,
instance_count=1,
base_job_name="abalone-xgboost-evaluation",
role=role,
sagemaker_session=pipeline_session,
)
evaluation_args = evaluation.run(
inputs=[
ProcessingInput(
source=model_s3_uri,
destination="/opt/ml/processing/model",
),
ProcessingInput(
source=step_preprocess.properties.ProcessingOutputConfig.Outputs["test"].S3Output.S3Uri,
destination="/opt/ml/processing/test",
),
],
outputs=[
ProcessingOutput(output_name="evaluation", source="/opt/ml/processing/evaluation"),
],
code="code/evaluation.py",
)
step_evaluation = ProcessingStep(
name="abalone-xgboost-evaluation",
step_args=evaluation_args,
depends_on=[step_notebook_job] # 明示的に紐づけ
)
ここで depends_on=[step_notebook_job] としている部分がポイントで、本来NotebookJobではなくTrainingJobを使う場合は、ここはTrainingJobのプロパティからモデルのS3 URIを取得するので、暗黙的な依存関係を読み取ってくれるのですが、NotebookJobの場合、プロパティにモデルのS3 URIが無いため、このS3 URIはパイプラインのパラメータとして定義して共通的に参照しています。よって依存関係は明示的に定義時に指定してあげる必要があります。
最後に定義したstepをパイプラインとしてまとめ、
from sagemaker.workflow.pipeline import Pipeline
pipeline_name = f"sample-notebook-job-pipeline"
# 作成
pipeline = Pipeline(
name=pipeline_name,
parameters=[
instance_type,
input_data,
model_s3_uri,
],
steps=[step_preprocess, step_notebook_job, step_evaluation]
)
パイプラインを更新します。
# 登録
pipeline.upsert(role_arn=role)
するとStudioのGUI画面からパイプラインを確認することができます。
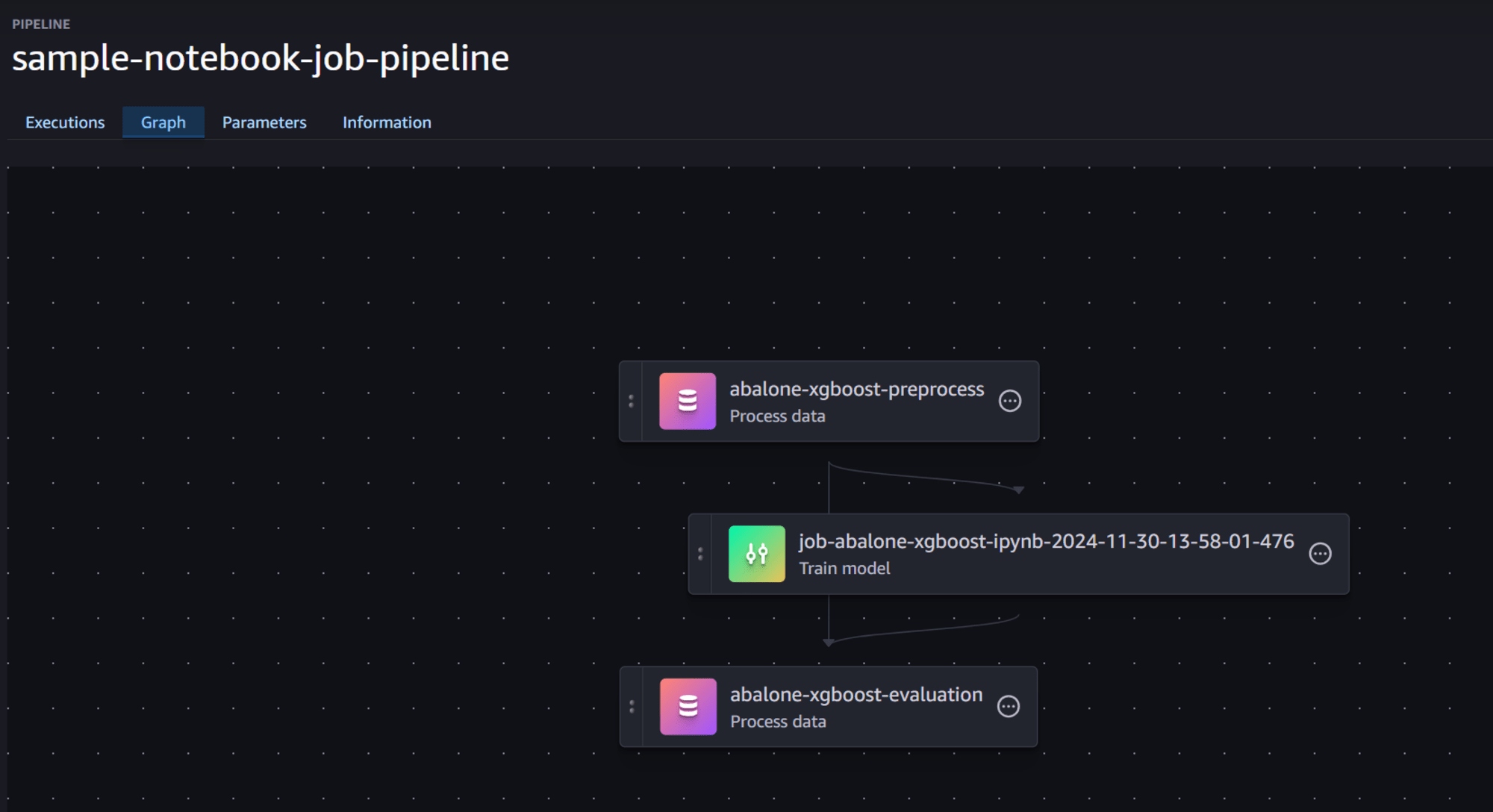
パイプラインの実行と処理待ち
パイプラインを実行して処理を待ちます。
# 実行
execution = pipeline.start()
# 完了待ち
execution.wait()
今回は15分以内で処理が完了する想定です。
実行結果の確認
pipelineの結果確認
以下で各ジョブのステータスを確認することができます。
execution.list_steps()
[{'StepName': 'abalone-xgboost-evaluation',
'StartTime': datetime.datetime(2024, 11, 30, 23, 6, 29, 136000, tzinfo=tzlocal()),
'EndTime': datetime.datetime(2024, 11, 30, 23, 11, 33, 629000, tzinfo=tzlocal()),
'StepStatus': 'Succeeded',
'Metadata': {'ProcessingJob': {'Arn': 'arn:aws:sagemaker:us-east-1:{アカウントID}:processing-job/pipelines-r0eqtmc006wi-abalone-xgboost-eval-UG1z9mWuyX'}},
'AttemptCount': 1},
{'StepName': 'job-abalone-xgboost-ipynb-2024-11-30-13-58-01-476',
'StartTime': datetime.datetime(2024, 11, 30, 23, 3, 19, 734000, tzinfo=tzlocal()),
'EndTime': datetime.datetime(2024, 11, 30, 23, 6, 28, 127000, tzinfo=tzlocal()),
'StepStatus': 'Succeeded',
'Metadata': {'TrainingJob': {'Arn': 'arn:aws:sagemaker:us-east-1:{アカウントID}:training-job/abalone-xgboost-notebook-job-r0eqtmc006wi-b7e42ekoxP'}},
'AttemptCount': 1},
{'StepName': 'abalone-xgboost-preprocess',
'StartTime': datetime.datetime(2024, 11, 30, 22, 58, 15, 515000, tzinfo=tzlocal()),
'EndTime': datetime.datetime(2024, 11, 30, 23, 3, 19, 36000, tzinfo=tzlocal()),
'StepStatus': 'Succeeded',
'Metadata': {'ProcessingJob': {'Arn': 'arn:aws:sagemaker:us-east-1:{アカウントID}:processing-job/pipelines-r0eqtmc006wi-abalone-xgboost-prep-HFWUFFsFWA'}},
'AttemptCount': 1}]
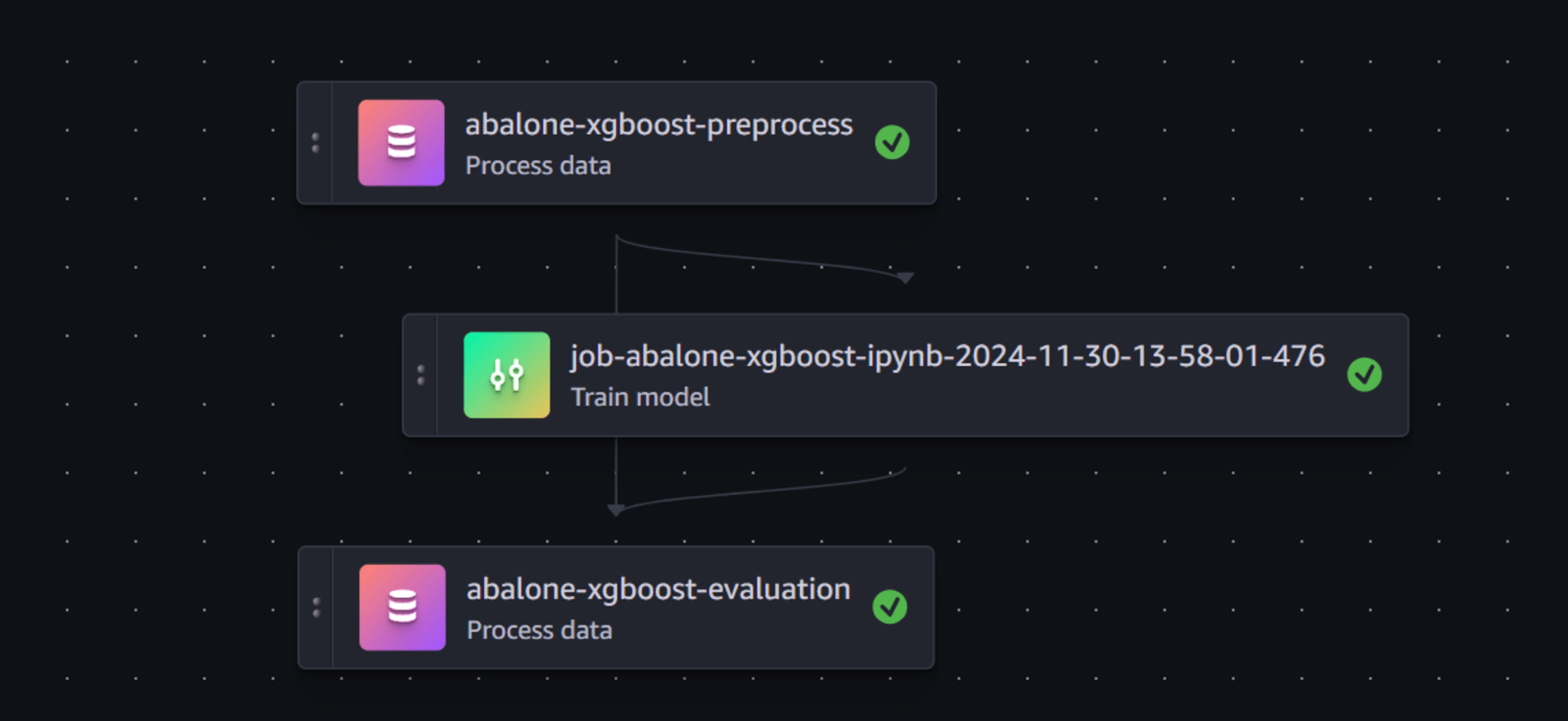
ProcessingJobとNotebookJob(実体はTrainingJob)で実行されたことが分かります。
またStudioの各実行結果の確認画面で、成功したかどうかを確認できます。
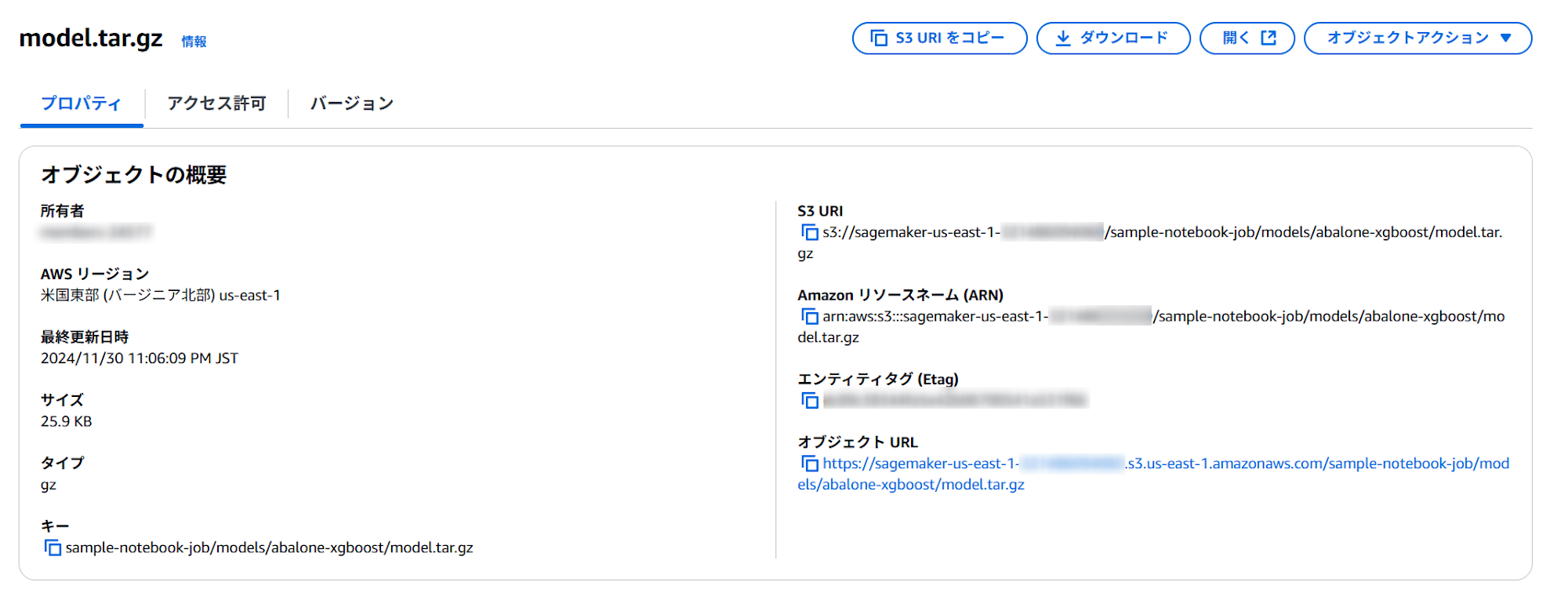
モデルの出力
モデルが以下に出力されていることを確認できました。
s3://{デフォルトバケット}/sample-notebook-job/models/abalone-xgboost/model.tar.gz
誤差評価結果の出力
誤差評価結果も以下に出力されていることを確認できました。
s3://{デフォルトバケット}/{パイプライン名}/{実行ID}/{ステップ名}/output/evaluation/evaluation.json
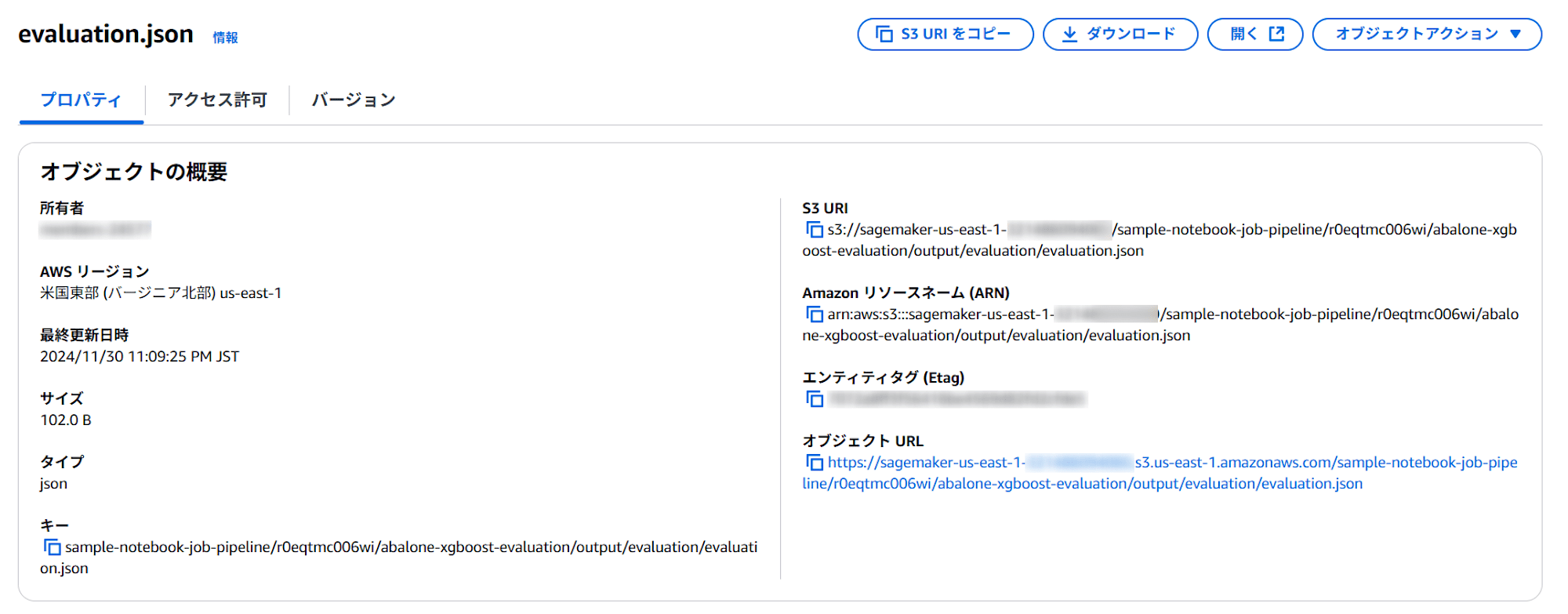
内容も正常に記録されているようです。
{"regression_metrics": {"mse": {"value": 4.262548012048034, "standard_deviation": 2.061813973658398}}}
まとめ
いかがでしたでしょうか。
前回はNotebook Job単体のpipelinesで検証しましたが、今回は前処理や誤差評価などと組み合わせてパイプラインを構成してみました。
これらをModel Registryへの登録やバッチ推論と組み合わせることも可能です。
今後そちらの紹介もしていきたいと思います。
本記事がご参考になれば幸いです。









